Table of Contents
Genehmigt
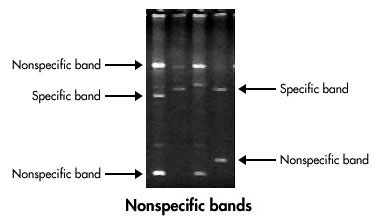
Wenn Sie auf Ihrem vertrauenswürdigen PC Fehlermeldungen zu pcr-Marketing und -Werbung sowie zur Fehlerbehebung sehen, sehen Sie sich diese Strategien zur Fehlerbehebung an.Viele der bevorzugten Probleme bei PCR und RT-PCR werden normalerweise während der Agarose-Gelelektrophorese gesehen, die die Reaktionsprodukte gezeigt hat. Dazu gehören unser Fehlen des erwarteten Erhaltungs- oder Amplifikationsprodukts, das Vorhandensein unspezifischer Produkte, übermäßiges Verschmieren und das Profil einer neuen Primer-Dimer-Bande.
Alte PCR diagnostiziert Probleme mit Ergebniskomponenten und, was noch wichtiger ist, Amplifikationsnetzwerken, indem sie das Gel laufen lässt. Verwandte Themen: PCR-Tools, PCR-Reagenzien, Entwicklung von PCR-Analysen und damit PCR-Optimierung, aber Assay.
Schritt nur eins – Denaturierung.Schritt 2 – Glühen.Die zweite Stufe ist die Expansion.Schritt viele – Elektrophoreseanalyse.
Arbeiten Sie in einem wirklich ausgewiesenen Bereich.PCR-Reagenzien und PCR-Produkte getrennt lagern.Aliquot.Bewahren Sie PCR-Röhrchen / -Punkte / -Racks separat auf.Ziehen Sie im Freien nicht den größten Teil der Folie vom Schlauch ab.Nutzen Sie einfach den Hauptmixer und fügen Sie das allgemeine Layout zuletzt hinzu.Trainiere andere.
Mascuch SJ, Fakhretaha-Aval S, Bowman JC, Ma MTH, Thomas G, Bommarius B, Ito C, Zhao L, Newnam GP, Matange KR, Thapa HR, Barlow B., Donegan RK, Nguyen NA, Saccuzzo EG, Obianyor KT, Karunakaran SK, P llet P., Rothschild-Mancinelli B., Mestre-Fos S., Gut-Metzler R., Bryksin AV, Petrov AS, Hazell M., Ibberson KB, Penev PI , Mannino R.G., Lam V.A., Garcia A.J., Kubanek D., Agarwal V., Hud N.V., Glass J.B., Williams L.D., Lieberman R.L. Maskuh S.J. et al. JBiolChem. 2020-11-13;295(46):15438-15453. doi: 10.1074/jbc.RA120.015434. Online veröffentlicht am 3. September 2020 JBiolChem. 2020 PMID: 32883809 Kostenloser PMC-Artikel.
PCR-Werbung erfordert ein feines Gleichgewicht zwischen der Art der Verstärkung spezifischer Produkte und der Bekämpfung der Produktion unspezifischer Heilmittel. Ziel dieser Studie könnte es gewesen sein, zu evaluieren, welche Parameter die Effizienz und Spezifität der DNA-Amplifikation einer Person beeinflussen.
Vermeiden Sie Sequenzprobleme.Primer-Homologie prüfen.Spiel für Anfänger T m.Ende mit G zusätzlich C.Vergessen Sie nicht, Abstandshalter zum Klonen/Zusammenbau von isothermen Restriktionsenzymen einzuschließen.Halten Sie eine eindeutig ausgewogene Primerkonzentration ein.
Mascuch SJ, Fakhretaha-Aval S, Bowman JC, Ma MTH, Thomas G, Bommarius B, Ito C, Zhao L, Newnam GP, Matange KR, Thapa HR, Barlow B., Donegan RK, Nguyen NA, Saccuzzo EG, Obianyor KT, Karunakaran SK, Pollet P., Rothschild-Mancinelli B., Mestre-Fos S., Gut-Metzler R., Bryksin AV, Petrov AS, Hazell M ., Ibberson KB, Penev PI, Mannino RG, Lam VA, Garcia AJ, Kubanek DM , Agarwal V., Khad N.V., Glass J.B., Williams L.D., Lieberman R.L. Maskuh S.J. de plus al. medRxiv. 1. September 2020: 2020.07.29.20163949. doi: 10.1101/2020.07.29.20163949. Bilden. medRxiv. 2020 PMID: 32766604 Kostenloser PMC-Artikel. Aktualisieren.
Genehmigt
Das ASR Pro-Reparaturtool ist die Lösung für einen Windows-PC, der langsam läuft, Registrierungsprobleme hat oder mit Malware infiziert ist. Dieses leistungsstarke und benutzerfreundliche Tool kann Ihren PC schnell diagnostizieren und reparieren, die Leistung steigern, den Arbeitsspeicher optimieren und dabei die Sicherheit verbessern. Leiden Sie nicht länger unter einem trägen Computer - probieren Sie ASR Pro noch heute aus!

Cold-Protocol Pring Harb; 2009; doi:10.1101/pdb.ip66
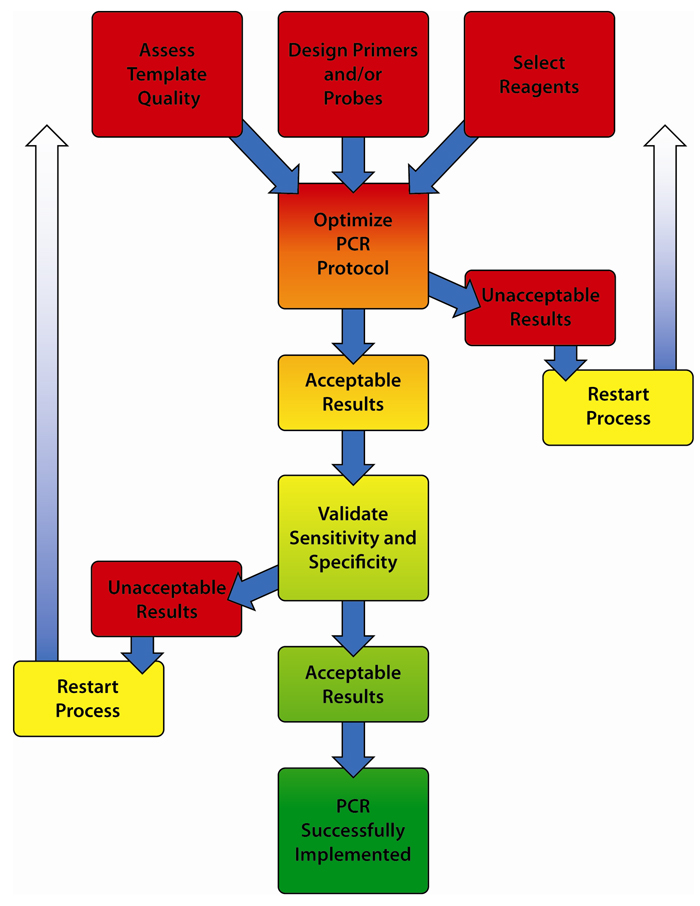
Die folgende Anleitung kann zur Fehlerbehebung bei PCR-Reaktionen verwendet werden. Verwenden Sie unseren Tm-Rechner, um mit den Begriffen zu experimentieren, und klicken Sie hier, um neue Sollwerte zu optimieren.
| Ansehen | Mögliche Ursache | Lösung |
|---|---|---|
| Sequenzfehler | Low-Excellence-Polymerase | |
| nicht optimale Bedingungen | ||
| Unausgeglichene Nukleotidkonzentrationen | ||
| Vorlagen-DNA war immer noch beschädigt | ||
| Die angeforderte Sequenz kann für ihren Host gefährlich sein | ||
| Ungültige Produktgröße | Falsche Vorwärmtemperatur | |
| Ladefehler | ||
| Falsche Mg-Konzentration | ||
| Nuklease-Kontamination | ||
| Keine Produkte | Falsche Vorwärmtemperatur | |
| Regierungswahn | ||
| Schlechte Grundierung, die spezifisch sein kann | ||
| Unzureichende Konzentration für Anfänger | ||
| Fehlende Antwortkomponente | ||
| Suboptimale Reaktionsbedingungen | ||
| Designvorlage von geringer Qualität | ||
| Vorhandensein des Inhibitors während der gesamten Reaktion | ||
| Nicht genügend Schleifen | ||
| Falsche Thermocycler-Programmierung | ||
| Variable Abschrecktemperatur | ||
| Kontamination von Impulsgefäßen oder Lösungen |
||
| Komplexes Modell |