Table of Contents
Approvato
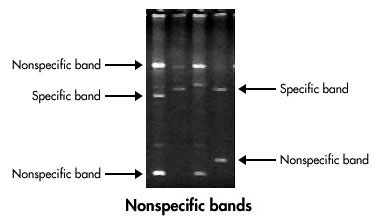
Se sul PC viene visualizzato un messaggio di errore di miglioramento e risoluzione dei problemi di pcr, dai un’occhiata a questi suggerimenti per la risoluzione dei problemi.Molti dei problemi preferiti con PCR e RT-PCR si trovano solitamente durante l’elettroforesi su gel di agarosio, che generalmente sono i prodotti di reazione. Questi includono l’eventuale assenza del mantenimento previsto per prodotto di amplificazione, la presenza che punta a prodotti non specifici, sbavature eccessive e le classifiche di una nuova banda primer-dimero.
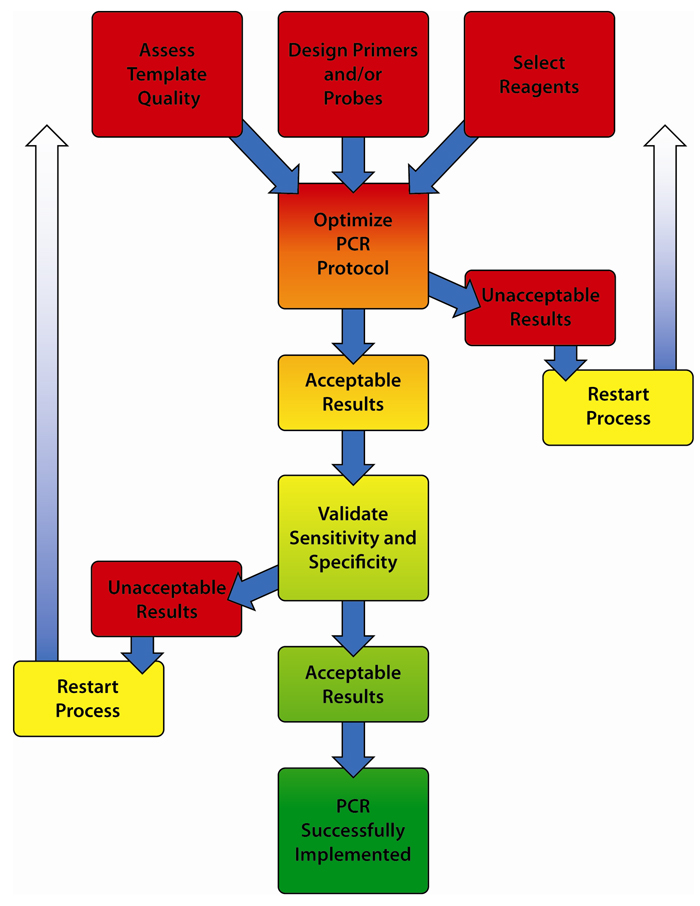
Ancient PCR diagnostica i problemi con il tipo di componenti di risposta e, soprattutto, le metodologie di amplificazione eseguendo il gel. Argomenti correlati: strumenti PCR, reagenti PCR, sviluppo di analisi PCR e quindi ottimizzazione PCR oltre al test.
Passo una persona specifica – Denaturazione.Passaggio 2: bagliore.La seconda fase è l’espansione.Fase quattro. – analisi elettroforesi.
Lavora in un’area completamente designata.Conservare separatamente i reagenti PCR ei prodotti PCR.Aliquota.Conservare separatamente le provette/punti/rastrelliere per PCR.Non staccare tutta la pellicola dal tubo all’aperto.Dipende solo dal mixer principale e aggiungi quel layout per ultimo.Allena gli altri.
Mascuch SJ, Fakhretaha-Aval S, Bowman JC, Ma MTH, Thomas G, Bommarius B, Ito C, Zhao L, Newnam GP, Matange KR, Thapa HR, Barlow B, Donegan RK, Nguyen NA, Saccuzzo EG, Obianyor KT, Karunakaran SK, P llet P., Rothschild-Mancinelli B., Mestre-Fos S., Gut-Metzler R., Bryksin AV, Petrov AS, Hazell M., Ibberson KB, Penev PI , Mannino RG, Lam V.A., Garcia A.J., Kubanek D. , Agarwal V., Hud N.V., Glass J.B., Williams L.D., Lieberman R.L. Maskuh SJ et al. JBiolChem. 2020-11-13;295(46):15438-15453. doi: 10.1074/jbc.RA120.015434. Pubblicato online il 3 settembre 2020 JBiolChem. 2020 PMID: 32883809 Articolo PMC gratuito.
Il marketing e la pubblicità della PCR richiedono un delicato equilibrio relativo alla sorta di amplificazione di prodotti specifici e alla lotta contro la produzione di integratori per la salute non specifici. Lo scopo di questo studio era di valutare quali parametri influenzano una nuova efficienza e specificità dell’amplificazione del DNA.
Evita problemi di sequenziamento.Controllare l’omologia del primer.Gioco a sostegno dei principianti T m.Termina con G in aggiunta C.Non dimenticare di includere distanziatori con clonazione/assemblaggio di enzimi di restrizione isotermici.Mantieni una favolosa concentrazione di primer bilanciata.
Mascuch SJ, Fakhretaha-Aval S, Bowman JC, Ma MTH, Thomas G, Bommarius B, Ito C, Zhao L, Newnam GP, Matange KR, Thapa HR, Barlow B, Donegan RK, Nguyen NA, Saccuzzo EG, Obianyor KT, Karunakaran SK, Pollet P., Rothschild-Mancinelli B., Mestre-Fos S., Gut-Metzler R., Bryksin AV, Petrov AS, Hazell M ., Ibberson KB, Penev PI, Mannino RG, Lam VA, Garcia AJ, Kubanek DM , Agarwal V., Khad N.V., Glass J.B., Williams L.D., Lieberman R.L. Maskuh SJ et al. medRxiv. 1 settembre 2020: 2020.07.29.20163949. doi: 10.1101/2020.07.29.20163949. Modulo. medRxiv. 2020 PMID: 32766604 Articolo PMC gratuito. Aggiornare.
Approvato
Lo strumento di riparazione ASR Pro è la soluzione per un PC Windows che funziona lentamente, presenta problemi di registro o è infetto da malware. Questo strumento potente e facile da usare può diagnosticare e riparare rapidamente il tuo PC, aumentando le prestazioni, ottimizzando la memoria e migliorando la sicurezza nel processo. Non soffrire più di un computer lento: prova ASR Pro oggi!

Protocollo freddo Pring Harb; 2009; doi:10.1101/pdb.ip66
È possibile utilizzare la seguente guida che può risolvere i problemi delle reazioni PCR. Usa il nostro calcolatore Tm per sperimentare gli approcci e fai clic qui per ottimizzare questi setpoint.
| Guarda | Possibile causa | Soluzione |
|---|---|---|
| Errore di sequenza | Polimerasi a bassa affidabilità | |
| condizioni non ottimali | ||
| Concentrazioni di nucleotidi sbilanciate | ||
| Il DNA del modello era ancora danneggiato | ||
| La sequenza essenziale potrebbe essere pericolosa per tutto l’ospite | ||
| Taglia prodotto non valida | Temperatura di preriscaldamento errata | |
| Errore di caricamento | ||
| Concentrazione di Mg errata | ||
| Contaminazione da nucleasi | ||
| Nessun prodotto | Temperatura di preriscaldamento errata | |
| Delusione del governo | ||
| Primer di pittura scadente che può essere specifico | ||
| Concentrazione di primer per pittura insufficiente | ||
| Componente verbale mancante | ||
| Condizioni di reazione non ottimali | ||
| Modello di design di bassa qualità | ||
| Presenza dell’inibitore durante tutta la tua reazione attuale | ||
| Cicli non adeguati | ||
| Programmazione del termociclatore errata | ||
| Temperatura di inibizione variabile | ||
| Contaminazione dei vasi impulsivi o soluzioni |
||
| Modello complesso |