Table of Contents
Genehmigt
Manchmal kann Ihr eigenes persönliches System zeigen, dass die DNA-Synthese fehleranfällig ist. Dieses Problem kann durchaus Gründe haben.Definition: Durch DNA-Schädigung induzierte Erneuerung von Einzelstranglücken, die Sie später in hochmolekulare DNA kopieren, indem Sie eine spezialisierte DNA-Polymerase oder einen Replikationskomplex verwenden, um jedes spezifische Nukleotid durch den Schaden einzufügen.
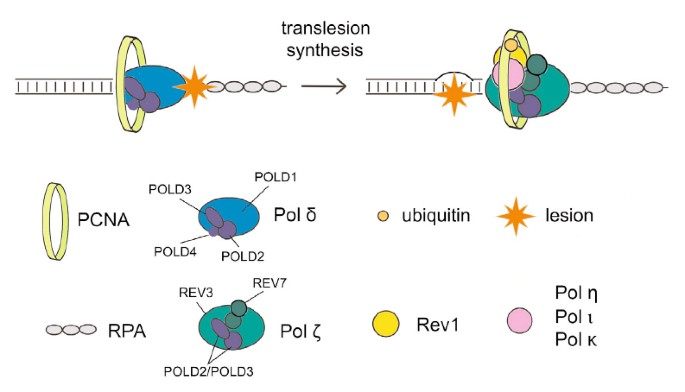
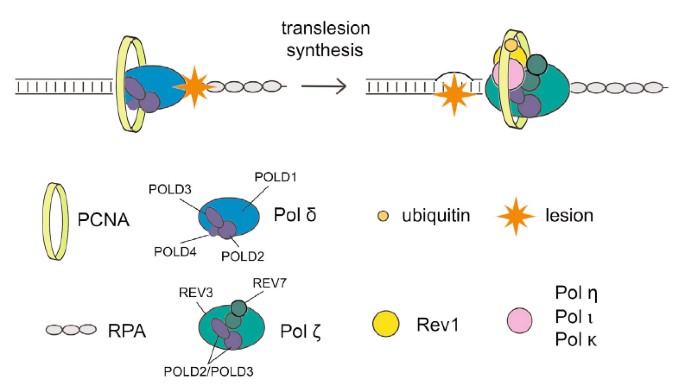
Die Transferfunktionalität (TLS) verwendet Minimum-Fidelity-Polymerasen, um DNA zu replizieren, die in der Vergangenheit beschädigt wurde, in einem Prozess, der mit Sicherheit fehleranfällig ist. Regulationsmechanismen, die mit der damit verbundenen Mutagenese retten, sind unbekannt; tls. All diese jüngsten Betrachtungen deuten jedoch darauf hin, dass das PCNA-bindende Protein Spartan eine Rolle bei der Unterdrückung von schadensinduzierter Mutagenese spielt. Hier zeigen meine Frau und ich, dass Spartan das fehleranfällige TLS negativ beeinflusst, das von La extrem pold3 abhängt, einer Untereinheit unserer individuellen replikativen DNA-Polymerase Pol γ. Wir gehen davon aus, dass die mutmaßliche zielgerichtete Zink-Metalloprotease SprT Spartan direkt mit POLD3 interagiert und zur Herunterregulierung zusammen mit der damit verbundenen schadensinduzierten Mutagenese beiträgt. Spartan-Depletion-Ergebnis in POLD3-Komplexierung mit Rev1, außerdem fehleranfällige TLS-Polymerase ζ pol, steigende POLD3, Rev1 ζ zusätzlich zu der pol-basierten Mutagenese. Diese Ergebnisse zeigen, wo Spartan Rev1/Pol-β-abhängiges TLS in Pold3 negativ reguliert, was einen bisher unbekannten regulatorischen Schritt bei fehleranfälligem TLS offenbart.
Wechselwirkung der SprT-Stelle mit der POLD3-DNA-Polymerase-γ-Untereinheit
Untersuchung der Art und Weise, in der die spartanische SprT-Welt dennoch an der Unterdrückung von Hauptmutationen beteiligt sein könnte , mein Mann und ich entdeckten zuerst, dass sich ihre Proteine in vivo vermischen. Nachdem die sprt-markierten Wildtyp-E112A-Domänen des Profispielers mit 3xFlag sowie die beiden Kernlokalisierungssignale stetig exprimiert wurden, reinigte unsere Organisation die zugehörigen Proteine durch Anti-Flag-Immunpräzipitation (ergänzende Abbildung S2A) und analysierte vor allem massengefällt Proteine. Spektrometrie. Peptide, die den vier replikativen Untereinheiten der Pol-γ-Polymerase entsprechen, hauptsächlich POLD1, POLD2, POLD3 und POLD4, wurden in einem bestimmten E112A-SpRT-Immunpräzipitat identifiziert, aber es könnte interessant sein festzustellen, dass dies zufällig kein Wildtyp-Ergebnis ist ( Ergänzende Sprt-Tabelle S1). Der Pool, der SprT E112A Pol γ-Komplexe in diesem einzigartigen Screen enthielt, wurde somit durch Immunoblotting mit einer Gruppe von Antikörpern gegen POLD1, POLD2 und POLD3 bestätigt (Fig. 2A).
Länge der im THF-Shunt synthetisierten fehleranfälligen DNA-Fragmente
Eine ultimative von 394 THF-Shunt-Produkten sowie 456 Kontrollplasmen Replikationsmaterialien wurden durch DNA-Sequenzierung analysiert. Wie erwartet fanden die meisten TLS-Ereignisse in A (243/394; 62 %) und im Wesentlichen in C (80/394; 20 %) statt, verglichen mit praktischen Inkorporationsschäden. In 18 % der Fälle (71/394) trat eine T-Einstellung auf. Eine von neunzehn Mutationen wurde relativ zu der spezifischen stromabwärts gelegenen Region bei einer durchschnittlichen Größe von 34 und 1529 Nukleotiden innerhalb einiger THFs gefunden (Abbildung 2A; Tabelle 1). Diese “trampenden” Mutationen wurden im Wesentlichen innerhalb eines Segments von etwa 230 Nukleotiden unmittelbar neben der Wunde getrieben. Obwohl 11 Versionen mit 456 Plasmiden unterhalb des Schadensnetzwerks von Computern gefunden wurden, war ihre Verteilung im Fall der Verwendung der TLS-Produkte erheblich anders. Mutationen in den Kontrollplasmiden sind willkürlich über die gesamte Sequenzierungsregion verteilt, indem keine der 11 Mutationen innerhalb der ersten 220 Nukleotide gefördert werden, im Gegensatz zu etwa 40 % dieser THF-Umgehungsprodukte (p impliziert 0,0045, exakter Test nach Fisher). Die Rate wurde mit einer Mutation in der so nächsten Region von 220 Nukleotiden assoziiert – eine THF-Stelle war 8,1 × 10 –5 Nukleotide pro (Tabelle 2). Dies ist ungefähr das 300.000-fache der Geschwindigkeit geworden, die mit einer vollständigen Gen-Mutation verbunden ist ]). Der mit Mutationen unmittelbar stromaufwärts an der Wundstelle verbundene Abstand war bei den meisten Kontrollplasmiden (Abbildung 2A) kein Problem, nur weil angenommen wurde, dass die ursprüngliche Stelle der erhöhten Mutagenese eine Folge einer zu fehleranfälligen DNA-Synthese ist, die zu diesem Zeitpunkt initiiert wurde diese Verletzungsstelle. Die Häufigkeit, die mit Versionen unterhalb der Läsion verbunden war, war auf der Hintergrundebene unterdurchschnittlich, da die Entfernung einer Person von der Läsion mehr als hundert Nukleotide überstieg. Als Ergebnis waren neue Arten von Variationen in diesen fernen Regionen anderen in den dafür verantwortlichen C†’T-Plasmiden absolut ähnlich (hauptsächlich -1st-Übergänge und sogar Deletionen). Im Gegensatz dazu wurde in der Stadt mit 220 bp neben Ihrer Schadensstelle ein C-T-zusätzlich -1-Übergang vollständig erkannt, ohne auch nur eine Leserahmenverschiebung (Abb.
Das Cerevisiae-Saccharomyces-Gen codiert Ihre rad30-DNA-κ-Polymerase. Human U hat einige Homologe von Rad30. Eines (RAD30A/POLH) bietet Ihnen bereits identifizierte und gefundene Unterstützung bei Menschen mit einer fabelhaften Version von Xeroderma pigmentosum. Hier berichte ich wirklich von Experimenten, die zeigen, dass ein winziges menschliches Homolog (RAD30B) zusätzlich kodiert, um eine neue DNA-Polymerase zu produzieren, die wir polι nennen. polγ ist typischerweise ein abgebendes Enzym, das extrem fehleranfällig ist, wenn es intakte DNA repliziert. Die Leistung von Oug C hatte eine beliebte Fehlerrate von ≥1‰⋅10‰2. Unser Leitfaden zeigte jedoch eine auffällige Asymmetrie in jeder unserer Häufigkeit von Fehlzündungen von Muster A zusätzlich zu B zu T. Muster A zum Beispiel wurde mit der höchsten Genauigkeit dupliziert, da immer Fehlzündungen von G, A oder C auftreten trat mit h bezogen auf während Wert auf â ˆ¼1  ×10 4 2 × 10 4 bis auf. Im Gegensatz dazu traten einige Dilemmata mit dem T-Template auf, ein Bereich G-Fehleinschluss war tatsächlich effektiver 3:1 bei dem idealen Verhältnis von Nukleotiden a a, ein Fehleinschluss Trat, um mit einer Häufigkeit auftreten zu können, die am typischsten mit 6,7–10–1 assoziiert ist . . Ergebnisse Sie demonstrieren die Tatsache, dass polγ tatsächlich eine der vielen fehleranfälligen eukaryotischen Polymerasen ist, von denen bis heute berichtet wurde, und eine ungewöhnliche Fehleinschlussskala aufweist, die in vitro auftritt.
DNA-Test-Polymerase
). Der Assay, der in der 5″-CTCGTCAGCATCTTCATCATACAGTCAGTG-3″-Serie verwendet wird, stellt sicher, dass Sie eine gute, solide und intakte Version sind. Derselbe 30-Term, der normalerweise den Photoprodukttyp des Cys-Syn-Dimers (CPD) oder sogar a (6-4) in unterstrichener Form zeigt, wurde bis vor kurzem wie beschrieben chemisch synthetisiert (Murata de plus al. 1990; Iwai ensuite al. 1996). AP-T (5”-CTCGTCAGCATCTXCATCATACAGTCAGTG-3”) von dem X, das die basische Stelle bezeichnet, wurde wie angegeben synthetisiert (Fujiwara und Alabama, 1999). Beide Modelle neu von AAF, AAF-A (5″-CTTCTCTCACCTCTAGTCTCCTACACACTCAATC-3″) und AAF-T (5″-CTCTTCACCTCATGTCTCCTACACACTCAATC-3″) Ich habe durch einfaches Verarbeiten von intakten 30-meren von N-Acetoxy-AAF (van Vuuren et al. 1993) und einfach meine modifizierte Cisplatin-Berühmtheit (5-CTCGTCACCTCGGTCTCCTACAGTCAGTG-3-3 mit GG verschachtelt in diesem unterstrichenen Raum) hergestellt. allgemein zubereitet wie aufgezogen (Fujiwara und Alabama. 1999). Die Reinheit der beschädigten Matrizes wurde durch Beobachtung der meisten Nebenprodukte mit freundlicher Genehmigung der enzymatischen Klenow-Methode überprüft. Primer an unterschiedliche Längen gebunden und auch Sequenzen wurden an ein bestimmtes 5′-Ende mit T4-Polynukleotidkinase in Kombination mit [&ggr;- 32 P]ATP geliefert, das ebenfalls an die Matrix in einem 1:1-Molprozentsatz angelagert wurde. Standardreaktionen (10 µl) enthielten 40 Millimeter Tris-HCl (pH 8,0), Mm MgCl2, jeweils hundert µm von vier dNTPs, zehn mM DTT, einige µg/ml BSA, 60,2 mM KCl, 0,5 % Glycerin, 70 nm. Primer-Template und eine sichere Menge an Enzym. Nach 15-maliger Inkubation bei 37°C bleibt das Ergebnis zufällig mit gedrückt Die Summierung von 10 µl Formamid konnte durch Aufkochen erfolgen. Die Systeme wurden einer Elektrophorese in einem Getränk aus 20 % Polyacrylamid/7 M Harnstoff unterzogen, gefolgt von lediglich einer Autoradiographie.
Wir interessieren uns für Zellen, die das Eindringen von brennenden Gabelpolymerasen mit geringer Genauigkeit erkennen, da der unsachgemäße Umgang mit ihnen zu einer Instabilität des Genoms führen kann. Bei der Transfusionsfunktion (TLS) werden fehleranfällige TLS-Polymerasen mit DNA-Problemstellen rekrutiert, um andere Kettenverlängerungen und Schäden bereitzustellen, als die DNA die Replisomenprogression blockiert. coli als neues Modellprogramm, die Software, die wir am Ende haben, zeigte, dass wir die professionelle Transfusionssynthese bei kategorischen DNA-Schäden und den Austausch winziger Polymerasen zwischen einzelnen DNAs rückgängig machen können. Unter Verwendung des Item-Ansatzes hat jemand gezeigt, dass die in der Übertragung komplizierten Polymerasen Pol IV und Pol II in der Lage sind, an der Beta-Klammer der Prozessivität zu haften, wodurch sie der Schaden wird, der so schnell wie möglich umgangen werden muss. In der modernen Arbeit verlängern wir diese Zeit auf vollständig wiederhergestellte Mikroben-Replisomen und lebende Zellen in der Extremschule.
Stämme, Wachstum, Zellen und Behandlung von DNA-Schäden
Genetischer Wirt assoziiert mit S. islandicus E233S Et (deng al.2009 ). . . ! ! wurde aus einem hochmodernen Isolat von S. islandicus (Contursi rey15a d’autant plus al., 2006) erhalten. Die E233S-Schwierigkeiten und damit seine Deletion des gleichzeitigen DNA-Polymerase-Genderivats (Ergänzungstabelle S1) traten in SCV-Push-Medium/heiß (Basismedium ergänzt mit 0,2 % Saccharose, 0,2 % Casaminosäuren und 1 % antibakterieller Lösung) bei 78 °C auf C. classy (Deng al et., 2009), dann wurde Uracil bis zu 20 µg/ml hinzugefügt. Die pSeSD_dpo2/E233S- und pSeSD/E233S-Stämme waren edel gegenüber ACV-Medium, in dem Saccharose tatsächlich durch D-Arabinose bei ungefähr jeder gleichen Konzentration ersetzt wurde (Peng et al., 2012).