Table of Contents
Aprovado
Às vezes, seu sistema pode exibir uma mensagem informando que a síntese de DNA é propensa a erros. Pode haver muitas razões para encontrar esse problema.Definição: Avivamento induzido por danos no DNA de lacunas de fita simples diretamente no DNA de peso molecular elevado após a cópia, obtendo uma polimerase de DNA especializada ou complexo de processamento para inserir cada nucleotídeo individual através do dano.
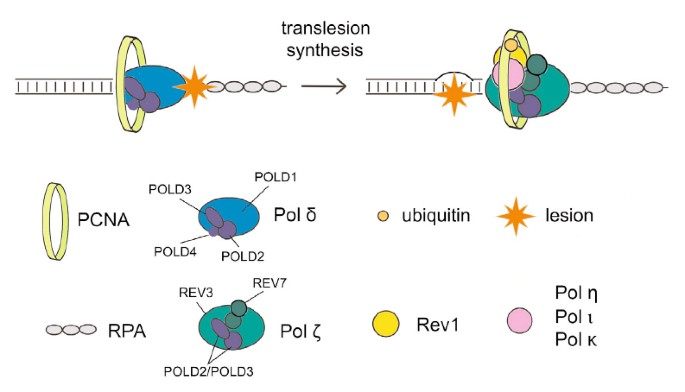
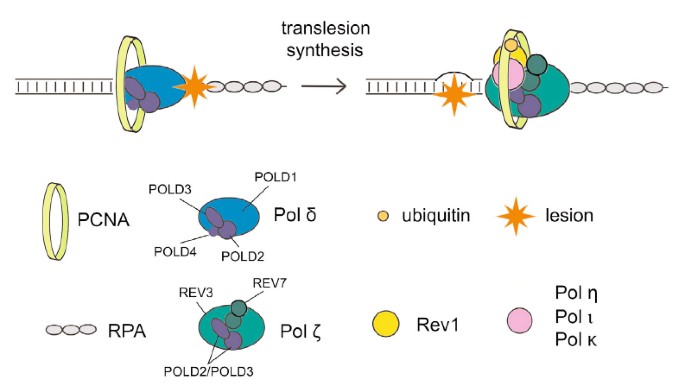
A execução de transferência (TLS) usa polimerases de baixa fidelidade para replicar o DNA que foi danificado no passado, no processo perfeito que é quase certo propenso a erros. Os mecanismos regulatórios que protegem a mutagênese com com são desconhecidos; tls. No entanto, quase todos esses estudos recentes sugerem que a proteína de ligação ao PCNA Spartan desempenha uma tarefa na supressão da mutagênese induzida por danos. Aqui, várias outras esposas e eu mostramos que o Spartan afeta negativamente o TLS propenso a erros, que depende do La extra pold3, uma importante subunidade de nossa DNA polimerase replicativa Pol γ. Nós postulamos que minha metaloprotease de zinco alvo putativa SprT Spartan em interage diretamente com POLD3 contribui para a regulação negativa de toda a mutagênese induzida por dano associada. A depleção espartana resulta na complexação de POLD3 com Rev1 e erro provável de polimerase de TLS ζ pol, aumentando a mutagênese de POLD3, Rev1 ζ e pol construída. Esses resultados indicam que o Spartan regula adversamente a função TLS dependente de Rev1/Pol-β em pold3, revelando um passo regulatório anteriormente não reconhecido em TLS propenso a erros.
Interação do local SprT com a subunidade γ da polimerase de DNA POLD3
Para estudar como o mundo SprT Spartan pode estar envolvido na repressão de grandes mutações, primeiro situamos que suas proteínas interagem enquanto in vivo. Depois que os domínios E112A de tipo selvagem marcados com pro sprt com 3xFlag e dois sinais de localização fischer foram expressos de forma estável, muitos de nossa organização purificaram as proteínas associadas em imunoprecipitação anti-Flag (Figura complementar S2A) e assim analisaram a maioria dos precipitados em massa peptídeos. espectrometria. Peptídeos correspondentes às 3 subunidades replicativas da Pol γ polimerase, em sua maioria POLD1, POLD2, POLD3 e POLD4, foram identificados no imunoprecipitado E112A SprT, mas é interessante quando você precisa notar que este não é um tipo selvagem real resultado (Tabela Sprt Suplementar S1). O conjunto contendo complexos SprT E112A Pol γ nesta tela foi primeiro assim confirmado por immunoblotting com um novo painel de anticorpos contra POLD1, POLD2 e POLD3 (Fig. 2A).
Comprimento de fragmentos de DNA propensos a erros sintetizados em derivação de THF
Um total por trás de 394 produtos de derivação de THF e 456 permanecem no topo do plasma produtos de replicação acabam analisados por sequenciamento de DNA. Conforme previsto, a maioria dos eventos de TLS ocorreu em A (243/394; 62%) e possivelmente C (80/394; 20%) em comparação com todos os problemas de incorporação. T-setting ocorreu em 18% das caixas (71/394). Uma das 18 mutações foi encontrada em relação ao downstream colocado a uma distância média de 33 e 1529 nucleotídeos de alguns THFs (Figura 2A; Tabela 1). Essas mutações de “carona” estavam essencialmente concentradas em um segmento importante de aproximadamente 220 nucleotídeos imediatamente adjacente à lesão. Embora nove versões tenham sido encontradas entre 456 plasmídeos alguns da rede de danos, sua circulação foi significativamente diferente quando se utilizou o tipo de em produtos TLS. Mutações nos plasmídeos de controle particulares são distribuídas aleatoriamente durante a região de sequenciamento, com nenhuma ligada às 11 mutações que se desenvolvem dentro de cada um dos nossos primeiros 220 nucleotídeos, em contraste o que será cerca de 40% dos subprodutos de THF (p = 0,0045, Fisher’s o próprio teste). A taxa foi associada agora com mutação na próxima região em 220 nucleotídeos – o local de informação do THF era de 8,1 x 10-5 nucleotídeos em cada (Tabela 2). Isso é 300.000 vezes mais rápido do que a geração completa. Muitas mutações no fermento de padeiro e certamente perto da taxa de erro classificada ao copiar DNA intacto trabalhando com Polζ purificado in vitro (5,6 × 10–4, [28 ]). A frequência associada a mutações imediatamente a montante do sítio ferido não diferiu de muitos plasmídeos de controle (Figura 2A) devido à ideia de que este sítio ligado ao aumento da mutagênese é devido à síntese de DNA propensa a erros iniciada nesta lesão relacionada ao exercício local. A frequência de versões na lesão foi baixa em nível de fundo, uma vez que sua distância da lesão ultrapassou dois mil nucleotídeos. Como resultado, os tipos que apontam para variações nessas regiões remotas são absolutamente semelhantes aos dos meus plasmídeos C†’T responsáveis por essas coisas (principalmente -1ª transições e deleções). Ao contrário, na região de duzentos e vinte bp adjacente ao dano relacionado ao local, uma mudança C-T e -1 foi completamente detectada sem um frameshift (Fig.
O gene Cerevisiae saccharomyces codifica a rad30 DNA-κ polimerase. Human U tem dois homólogos semelhantes a Rad30. Um (RAD30A/POLH) já foi identificado e não está funcionando em humanos com uma versão ligada ao xeroderma pigmentoso. Aqui relatamos ensaios mostrando que um segundo homólogo real (RAD30B) codifica adicionalmente uma nova DNA polimerase que chamamos de polι. polγ é geralmente uma enzima de entrega que é extremamente propensa a erros ao duplicar DNA intacto. A precisão Oug C teve uma taxa de erro típica de ≥1‰⋅10‰2. No entanto, nossa pesquisa revelou uma grande assimetria impressionante na frequência de falha de disparo do padrão A na adição de B a T. O padrão A, por exemplo, foi reproduzido com inquestionavelmente a mais alta fidelidade, assim como a falha de disparo G, A ou C, que se materializou com h relacionado por valor para que você possa ˆ¼1  ×10 4 2 × 10 4 bis. Pelo contrário, alguns erros ocorreram acompanhados pelo modelo T, em algum lugar G erro de inclusão era realmente mais preferível 3:1 na proporção ideal de nucleotídeos um enorme a, erro de inclusão Trat t ocorreu com uma frequência de 6,7-10-1. . Resultados Eles demonstram que polγ é com certeza uma das polimerases eucarióticas mais propensas a erros relatadas até o momento e desenvolveu um espectro incomum de inclusão incorreta que chega lá in vitro.
Teste de DNA Polimerase
). O ensaio usado com a nossa própria série 5″-CTCGTCAGCATCTTCATCATACAGTCAGTG-3″ mostra ser cada modelo bom, sólido e intacto. O termo realmente 30 mostrando o tipo de fotoproduto envolvido com o dímero Cys-Syn (CPD) ou um (6-4) sublinhado mnaturalmente, foi produzido quimicamente como descrito anteriormente (Murata de ou mais al. 1990; Iwai et al. 1996 ). AP-T (5”-CTCGTCAGCATCTXCATCATACAGTCAGTG-3”) a partir do X exacto que denota o local de base foi de facto sintetizado como descrito (Fujiwara e Alabama. 1999). Ambos os modelos modificados por AAF, AAF-A (5″-CTTCTCTCACCTCTAGTCTCCTACACACTCAATC-3″) e AAF-T (5″-CTCTTCACCTCATGTCTCCTACACACTCAATC-3″) Eu preparei processando 30-mers não danificados de N-acetoxi-AAF como escrito (van Vuuren et al. 1993) e modelo de cisplatina modificado privado (5-CTCGTCACCTCGGTCTCCTACAGTCAGTG-3-3 com GG aninhado no espaço sublinhado). mais comumente preparado como descrito (Fujiwara e Alabama. 1999). A pureza das matrizes danificadas foi verificada observando em grande parte os subprodutos por todo o método enzimático de Klenow. primers limitados a diferentes comprimentos e sequências foram originalmente entregues à extremidade 5′ na polinucleotídeo quinase de T4 e [γ-32P]ATP, ainda recozidos à matriz em uma porcentagem molar de 1:1. Os efeitos padrão (10 µl) continham 40 mM de Tris-HCl (pH 8,0), Mm MgCl2, 100 µm apenas cerca de quatro dNTPs, 10 mM DTT, alguns µg/ml de BSA, 60,2 milímetros KCl, 0,5% de glicerina, 60 nm. modelo de primer de tinta e uma certa quantidade com enzima. Após 15 minutos relativos à incubação a 37°C, a reação permanecePressionada com pouca reflexão com Soma de dez μl de formamida é realizada por fervura. Os produtos foram submetidos a eletroforese em poliacrilamida líquida 20%/ureia 7M seguida de autorradiografia.
Podemos estar interessados em como as células determinam a penetração de polimerases de divisão de replicação com baixa precisão, uma vez que a manutenção inadequada delas leva a diferenças no genoma. Na síntese de transfusão (TLS), as polimerases TLS propensas a erros são recrutadas para os locais de ocorrência do DNA para fornecer mais alongamento da corda, dano do que o DNA que fragmenta a progressão do replissoma. coli como um programa de TV modelo, o software que mostramos que parece que podemos fazer engenharia reversa da combinação de transfusão em danos de DNA específicos para o local, além da troca de polimerases em algum lugar entre DNAs individuais. Usando essa abordagem, o homem ou a mulher mostraram que a Pol IV também as polimerases Pol II envolvidas na transmissão de televisão são capazes de vincular o experimento com grampo de processividade, permitindo que a lesão seja contornada rapidamente. No trabalho diário existente, estendemos esse tempo para replisomes microbianos totalmente restaurados e células da vida diária no ensino médio.
cepas, crescimento, células e tratamento de danos no DNA
hospedeiro genético associado a S. islandicus E233S Et (deng al.2009 ). . . parece ter sido obtido a partir de um isolado inovador conectado a S. islandicus (Contursi rey15a et alabama., 2006). A cepa E233S e ergo sua deleção de cada derivado do gene da DNA polimerase (Tabela Suplementar S1) parecia estar em meio push SCV (base pequena suplementada com 0,2% de sacarose, 0,2% de ácidos graxos casamino e 1% de solução desinfetante) dentro da temperatura de 78°C C. cultivada (Deng al ainsi que., 2009), depois foi adicionado uracil e 20 µg/mL. As cepas pSeSD_dpo2/E233S e, além disso, pSeSD/E233S foram nobres na estratégia ACV em que a sacarose foi substituída e também pela D-arabinose aproximadamente no mesmo reagrupamento (Peng et al., 2012).