Table of Contents
Aprobado
A veces, su sistema puede mostrar un mensaje que indica que la síntesis de ADN sería propensa a errores. También puede haber muchas razones para este gran problema.Definición: renovación inducida por daños en el ADN junto con brechas de una sola hebra directamente en exceso de ADN de alto peso molecular después de copiar usando una polimerasa de ADN especializada o un complejo de replicación, puede insertar cada nucleótido específico a través de todo el daño.
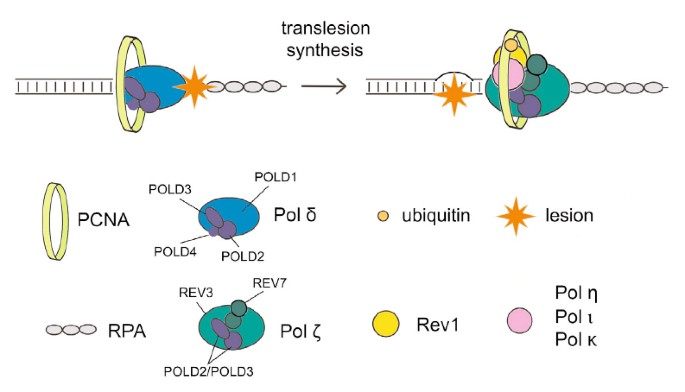
Interacción del sitio SprT con la subunidad γ de la ADN polimerasa POLD3
Para estudiar cómo la arena SprT Spartan puede estar involucrada en la principal represión de mutaciones, primero descubrimos que las proteínas individuales interactúan in vivo. Después de que cada dominio E112A de tipo salvaje etiquetado pro sprt completo con 3xFlag y dos signos de advertencia de localización nuclear se expresaron de manera estable, nuestra organización purificó las proteínas asociadas mediante inmunoprecipitación anti-Flag (Figura complementaria S2A) y analizó una gran cantidad de la masa- proteínas precipitadas. espectrometría Los péptidos correspondientes a las cuatro subunidades replicativas de la polimerasa Pol γ, principalmente POLD1, POLD2, POLD3 y POLD4, se mencionaron en el inmunoprecipitado E112A SprT, más bien es interesante notar que a menudo este no es un resultado final de tipo salvaje (Supplementary Sprt Tabla S1). La natación que contenía los edificios SprT E112A Pol γ en esta pantalla se validó por inmunotransferencia con un panel similar a los anticuerpos contra POLD1, POLD2 y POLD3 (Fig. 2A).
Longitud de los fragmentos de ADN propensos a errores sintetizados en derivación THF
Un total de 394 productos de derivación THF y 456 productos de imitación de plasma de control fueron analizado mediante secuenciación de ADN. Como era de esperar, la mayoría de los eventos de TLS ocurrieron en A (243/394; 62 %) y, además, posiblemente en C (80/394; 20 %) en comparación con todos los daños por incorporación. Engaste en T formado en el 18% de los casos (71/394). Una de las 18 mutaciones se encontró en la región aguas abajo a una distancia promedio de 34 y 1529 nucleótidos de algunos THF (Figura 2A; Tabla 1). Estas mutaciones de “autostop” se concentraron esencialmente en un segmento proveniente de aproximadamente 220 nucleótidos inmediatamente adyacentes a la lesión. Aunque se encuentran 11 versiones entre 456 plásmidos debajo de alguna red dañada, su distribución fue dramáticamente diferente cuando se usaba en productos TLS. Las mutaciones en los plásmidos de control se distribuyen aleatoriamente a lo largo de una región de secuenciación, y ninguna de las 17 mutaciones se desarrolla dentro de los primeros 230 nucleótidos, en contraste con casi el 40 % de los dispositivos de derivación de THF (p = 0,0045, prueba exacta de Fisher). La tasa se asoció con la mutación con respecto a la siguiente región de 230 nucleótidos – el sitio THF fue de 8,1 x 10-5 nucleótidos por (Tabla 2). Esto es alrededor de 300 000 veces mayor que la velocidad de un gen completo. Muchas mutaciones en la levadura de panadería y casi con toda certeza sellaron la tasa de error indicada al copiar el ADN intacto con Polζ purificado in vitro (5,6 × 10–4, [28]). La frecuencia asociada con las mutaciones apropiadas aguas arriba del sitio de la herida funcionó como un chef no difiere de la mayoría de los plásmidos manipulados (Figura 2A) debido a la recomendación de que este sitio de mutagénesis creciente se debe a un ejercicio de ADN propenso a errores iniciado en este sitio de la lesión. La frecuencia de versiones debajo de la llaga fue baja a la altura del fondo, ya que su distancia a la llaga excedía los doscientos nucleótidos. Como resultado, los tipos de cambios en estas regiones remotas fueron absolutamente iguales a los de los plásmidos C†’T responsables de ellos (principalmente cambios y eliminaciones -1st). Por el contrario, en la última región de 220 pb contigua al sitio del daño, se detectó bastante una transición C-T y -1 sin un cambio de marco de lectura (Fig.
El gen de Cerevisiae saccharomyces codifica su ADN-κ polimerasa rad30. Human U tiene dos homólogos de Rad30. Ya se ha identificado uno (RAD30A/POLH) y además se ha encontrado que es defectuoso en personas con una versión de xeroderma pigmentoso. Aquí informamos experimentos que muestran un segundo código químico homólogo humano (RAD30B) para una nueva ADN polimerasa que llamamos polι. polγ es comúnmente una enzima de entrega que es severamente propensa a errores cuando se replica ADN intacto. El rendimiento de Oug C tuvo una buena tasa de error típica sólida de ≥1‰⋅10‰2. Sin embargo, nuestra investigación reveló una asimetría deslumbrante en la frecuencia de fallas relacionadas con el patrón A además de B a T. El patrón A, solo para ilustrar, se reprodujo con la mayor fidelidad, al igual que las fallas de G, A o C, lo cual ocurrió con s relacionado por valor a â ˆ¼1  ×10 4 2 × 10 4 bis. Por el contrario, se produjeron algunos errores con la plantilla T, en algún lugar la inclusión errónea de G era realmente preferible 3:1 en la proporción correcta de nucleótidos a a, la inclusión errónea Trat t se produjo con una consistencia de 6,7–10–1. . Resultados Ellos corroboran que polγ es de hecho una de las polimerasas eucariotas más propensas a errores reveladas hasta la fecha y exhibe un espectro de inclusión errónea inesperado que ocurre a través de vitro.
Prueba de ADN polimerasa
). El análisis utilizado con la serie 5″-CTCGTCAGCATCTTCATCATACAGTCAGTG-3″ puede demostrar ser un modelo bueno, potente e intacto. El mismo término de 30 que implica el tipo de fotoproducto del dímero Cys-Syn (CPD) o (6-4) cuando se trata de subrayado mnaturalmente, se sintetizó químicamente como se explicó anteriormente (Murata de plus al. 1990; Iwai et al. 1996). AP-T (5”-CTCGTCAGCATCTXCATCATACAGTCAGTG-3”) a partir de la X que indica el sitio base se sintetizó desde que se describió (Fujiwara y Alabama, 1999). Ambos modelos modificados por AAF, AAF-A (5″-CTTCTCTCACCTCTAGTCTCCTACACACTCAATC-3″) y AAF-T (5″-CTCTTCACCTCATGTCTCCTACACACTCAATC-3″) Preparé procesando 30-mers intactos con respecto a N-acetoxi-AAF como se indicó (van Vuuren avec al. 1993) y mi modelo de cisplatino mejorado (5-CTCGTCACCTCGGTCTCCTACAGTCAGTG-3-3 con GG anidado operando en el espacio subrayado). generalmente preparado incluso como se describe (Fujiwara y Alabama. 1999). La pureza de las matrices dañadas tuvo que comprobarse observando la mayoría de sus subproductos por el camino enzimático de Klenow. cebadores unidos a múltiples longitudes y secuencias se entregaron para permitirles el extremo 5′ con polinucleótido quinasa T4 y [γ-32P]ATP, también hibridados con la matriz en un porcentaje molar de 1:1. Las reacciones estándar (10 µl) contenían de forma segura Tris-HCl 40 mM (pH 8,0), MgCl2 Mm, 100 µm de cada uno de numerosos dNTP, DTT 10 mM, una cantidad baja de µg/ml BSA, KCl 60,2 mM, glicerina al 0,5 %, 60 Nuevo Méjico. cebador plantilla por lo que una cierta cantidad de enzima. Después de 15 minutos de incubación cerca de los 37°C, la reacción queda Presionada al azar con La suma de 10 μl involucrados con formamida se realiza por cocción. Los productos se sometieron a electroforesis en poliacrilamida líquida al 20%/urea 7 M seguida de autorradiografía.
Cepas, crecimiento, células y tratamiento del daño del ADN
Huésped genético relacionado con S. islandicus E233S Et (deng al.2009 ). . . se obtuvo proporcionado por un aislamiento innovador de S. islandicus (Contursi rey15a et al., 2006). La cepa E233S y, por lo tanto, su erradicación de cada marca del gen de la ADN polimerasa (Tabla complementaria S1) apareció en medio de empuje SCV (medio base suplementado y sacarosa al 0,2 %, casaminoácidos al 0,2 % en una solución desinfectante al 1 %) a 78 °C C. cultivada ( Deng al et., 2009), posteriormente se añadió uracilo a 31 µg/mL. Las cepas pSeSD_dpo2/E233S y pSeSD/E233S eran originalmente nobles en medio ACV en este también se reemplazó la sacarosa por D-arabinosa que se encuentra aproximadamente en la misma concentración (Peng ensuite al., 2012).