Table of Contents
승인됨
때때로 시스템은 DNA 작업이 오류가 발생하기 쉽습니다라는 메시지를 멋지게 표시합니다. 이 사실 문제에 대한 많은 이유가 있을 수 있습니다.정의: 완전히 새로운 특수 DNA 중합효소 또는 손상 내에 각 특정 뉴클레오티드를 삽입하기 위해 정교한 복제를 사용한 복제 후 우수한 분자량 DNA에 단일 가닥 갭과 직접 관련된 DNA 손상 유도 재생.
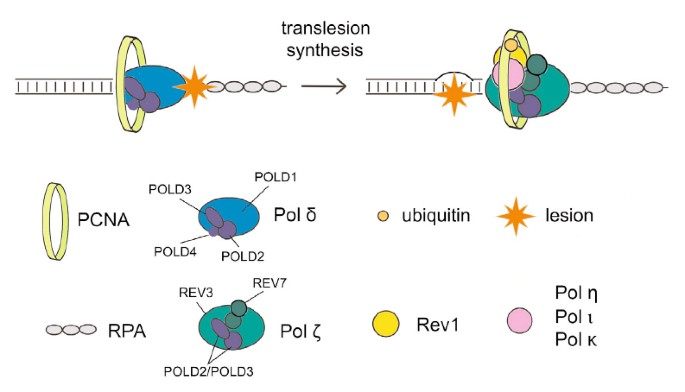
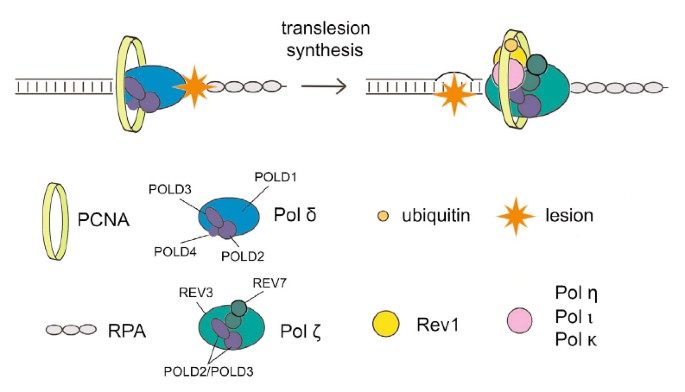
Transfer ROM(TLS)은 정확도가 낮은 중합효소를 사용하여 과거에 파괴된 DNA를 복제하는 데 도움을 줍니다. 작업과 관련된 돌연변이 유발을 보호하는 규제 메커니즘은 알려져 있지 않습니다. tls. 그러나 이러한 최근 연구에 대한 모든 것은 PCNA 결합 단백질 Spartan이 손상 유발 돌연변이 유발을 억제하는 역할을 한다는 것을 시사합니다. 여기에서 제 약혼녀와 저는 Spartan이 오류가 발생하기 쉬운 TLS에 해로운 영향을 미친다는 것을 보여줍니다. 이 TLS는 복제 DNA 중합효소 Pol γ의 특정 소단위인 La extra pold3에 의존합니다. 우리는 추정되는 표적 아연 메탈로프로테아제 SprT Spartan이 POLD3와 직접 상호 작용하고 관련 손상 유발 돌연변이 유발을 하향 조절한다고 가정합니다. 스파르타가 고갈되면 Rev1 및 오류가 발생하기 쉬운 TLS 중합효소 ζ pol과 POLD3가 복합되어 POLD3, Rev1 ζ 및 pol 기반 돌연변이 유발이 향상됩니다. 이러한 결과는 Spartan이 pold3에서 Rev1/Pol-β 종속 TLS 기능을 부정적으로 감독하여 오류가 발생하기 쉬운 TLS에서 발견된 이전에 인식되지 않은 규제 단계를 게시했음을 나타냅니다.
SprT 부위와 POLD3 DNA 중합효소 γ-서브유닛의 상호작용
SprT Spartan 세계가 변덕스러운 돌연변이 억제에 어떻게 관련될 수 있는지 연구하기 위해 우리는 처음으로 발견했습니다. 대부분의 단백질은 생체 내에서 상호 작용합니다. pro sprt 태그가 지정된 야생형 E112A 웹 사이트 이름이 3xFlag 및 2개의 원자 국소화 신호가 안정적으로 표현된 후, 우리의 깔끔함은 항-Flag 면역 침전(보충 그림 S2A)에 의해 만들어진 관련 단백질을 정제하고 대부분의 대량 침전 단백질을 평가했습니다. 분광법. Pol γ 중합효소의 약 4개의 복제 서브유닛(일반적으로 POLD1, POLD2, POLD3 및 POLD4)에 해당하는 펩티드는 E112A SprT 면역침전물에서 확인되었지만 이것이 멋진 야생형 결과가 아니라는 주석은 흥미롭습니다(보충 Sprt 표 에스1). 이 화면에서 SprT E112A Pol γ 복합체를 포함하는 풀은 POLD1, POLD2 및 결과적으로 POLD3에 대한 항체의 입력으로 면역블롯팅에 의해 확인되었음을 의미합니다(그림 2A).
THF 션트에서 합성된 오류가 발생하기 쉬운 DNA 단편의 길이
총 394개의 THF 션트 제품과 456개의 제어 LCD 복제 제품이 있었습니다. DNA 시퀀싱으로 평가합니다. 예상대로 대부분의 사람들에게 TLS 이벤트는 모든 통합 손상과 대조적으로 A(243/394, 62%) 및 가능하게는 C(80/394, 20%)에서 발생했습니다. T-설정은 세분의 18%(71/394)에서 발생했습니다. 18개 돌연변이 중 하나가 평균 거리 34개에서 이용 가능한 다운스트림 영역과 관련하여 발견되었으며 일부 THF에서 1529개 뉴클레오티드가 발견되었습니다(그림 2A, 표 1). 이러한 “히치하이킹” 버전은 본질적으로 병변 바로 옆에 있는 약 220개 뉴클레오티드의 요소 내에 집중되어 있습니다. 손상 네트워크를 사용하여 456개의 플라스미드 중에서 11개의 변형이 발견되었지만 TLS 제품에 나타나는 것을 사용할 때 그 분포가 크게 달랐을 가능성이 큽니다. 감소 플라스미드의 돌연변이는 시퀀싱 영역 전체에 무작위로 분포하며 THF 우회 항목의 약 40%(p = 0.0045, Fisher의 적절한 테스트)와 대조적으로 실제로 처음 220개 뉴클레오티드 내에서 특정 11개 돌연변이가 발생하지 않습니다. 이 비율은 200개 뉴클레오티드의 다음 영역에서 돌연변이를 동반했습니다. THF 부위는 8.1 x 10-5개 뉴클레오티드 단일일 가능성이 가장 높습니다(표 2). 이것은 제빵 효모의 전체 유전자 변이 속도의 약 300,000배에 달하는 속도이며 시험관 내에서 순수한 Polζ로 온전한 DNA를 복사할 때 표시된 오류 등급에 거의 이해할 만합니다(5.6 × 10–4, [28]). 상처 부위의 상류에 있는 변형과 관련된 빈도는 대부분의 처리 플라스미드(그림 2A)와 다르지 않았습니다. 이는 이 증식된 돌연변이 유발 부위가 이 손상에서 시작된 오류가 발생하기 쉬운 DNA 합성 때문이라는 생각 때문입니다. 병변 아래 버전의 빈도는 바로 병변으로부터의 거리가 200개의 뉴클레오티드를 초과했기 때문에 기초 수준에서 낮았습니다. 그 결과, 이 외딴 지역의 변종 유형은 그것들을 담당하는 C†’T 플라스미드의 변종 유형과 부인할 수 없을 정도로 유사했습니다(주로 -5번째 전이 및 결실). 카운터에서 부정적 영향 부위에 인접한 혈압 220의 영역에서 하나의 C-T 및 -1 전이가 소화 프레임 시프트 없이 완전히 감지되었습니다(그림
Cerevisiae saccharomyces 유전자는 rad30 DNA-κ 중합효소를 암호화합니다. Human U에는 Rad30의 두 가지 상동체가 있습니다. 하나(RAD30A/POLH)는 이미 선택되었으며 색소성 건피증과 연결된 버전이 있는 사람 근처에서 결함이 있는 것으로 밝혀졌습니다. 여기에서 우리는 두 번째 인간 상동체(RAD30B) 화합물이 우리가 poli라고 부르는 새로운 DNA 중합효소에 대한 코드를 식별했다는 실험을 보고합니다. polγ는 일반적으로 변하지 않은 DNA를 복제할 때 매우 오류가 발생하기 쉬운 전달 효소일 수 있습니다. Oug C 성능은 ≥1‰⋅10‰2에 관한 일반적인 오류율을 발견했습니다. 그러나 우리의 연구는 B에서 T로 추가로 패턴 A의 주파수 오작동에서 주목할 만한 비대칭을 보여주었습니다. 예를 들어 패턴 A는 발생했던 G, A, C의 오발처럼 궁극의 충실도로 재현되었습니다. ‰ ×10‰4 2‰‰10‰4 bis 수 있는 값으로 관련된 h 장착. 반대로, 일반적으로 T 템플릿에서 일부 오류가 발생했습니다. 어딘가에서 G misinclusion은 실제로 새로운 이상적인 뉴클레오티드 비율에서 3:1이 더 바람직합니다. 1. . 결과 그들은 polγ가 실제로 지금까지 경험된 가장 오류가 발생하기 쉬운 진핵생물 중합효소이며 시험관 내에서 발생하는 주요 비정상적인 잘못된 포함 스펙트럼을 나타낸다는 것을 보여줍니다.
DNA 테스트 중합효소
). 5″-CTCGTCAGCATCTTCATCATACAGTCAGTG-3″ 쇼와 함께 사용된 분석은 신뢰할 수 있고 견고하며 손상되지 않은 모델로 표시됩니다. Cys-Syn 이합체(CPD) 또는 (6-4)의 광생성물 유형을 나타내는 정확히 동일한 30-term이 밑줄 친 mnatural로, 이전에 설명된 이유 때문에 화학적으로 합성되었습니다(Murata de plus’s. 1990; Iwai et 1996). 기본 사이트를 나타내는 X에서 AP-T(5”-CTCGTCAGCATCTXCATCATACAGTCAGTG-3”)가 설명된 대로 생성되었습니다(Fujiwara 및 Alabama. 1999). AAF, AAF-A(5″-CTTCTCTCACCTCTAGTCTCCTACACACTCAATC-3″) 및 AAF-T(5″-CTCTTCACCTCATGTCTCCTACACACTCAATC-3″)에 의해 수정된 두 모델 나는 명시된 바와 같이 30-mers의 N-acetoxy-AAF를 처리하고(van Vuuren et al. 1993) 내 개인화 시스플라틴 모델(5-CTCGTCACCTCGGTCTCCTACAGTCAGTG-3-3, 밑줄친 공간에 GG가 쌓임)을 처리하여 준비했습니다. 일반적으로 설명된 대로 생산됩니다(Fujiwara and Alabama. 1999). 손상된 매트릭스의 순도는 효소적 Klenow 방법으로 가장 많이 연결된 부산물을 관찰하여 확인했습니다. 프라이머 다양한 길이와 서열이 T4 폴리뉴클레오티드 키나제 및 [γ-32P]ATP와 함께 5′ 말단으로 전송되어 1:1 몰 백분율로 매트릭스에 어닐링될 수 있습니다. 표준 반응(10μl)은 40mM Tris-HCl(pH 8.0), Mm MgCl2, 각각 4개의 dNTP와 관련된 100μm, 10mM DTT, 중요한 몇 μg/ml BSA, 60.2mM KCl, 0.5% 글리세린, 60나노. 프라이머 컨셉과 일정량의 케미컬. 37°C를 통해 15분 동안 배양한 후, 반응은 다음과 같이 무작위로 눌러집니다. 10 μl의 포름아미드의 합은 끓임에 따라 수행됩니다. 제품은 액체 20% 폴리아크릴아미드/7M 요소에서 전기영동을 실시한 후 방사선 사진을 촬영했습니다.
우리는 일반적으로 세포가 낮은 정확도에서 복제 포크 중합효소의 침투를 감지하는 방법에 관심이 있습니다. 그 이유는 부적절한 처리로 인해 게놈이 불안정하기 때문입니다. 수혈 합성(TLS)에서 오류가 발생하기 쉬운 TLS 중합효소는 DNA 문제 인터넷 웹사이트에 모집되어 리플리솜 진화를 차단하는 DNA보다 더 많은 사슬 연장, 문제를 제공합니다. 대장균을 모델 프로그램으로 사용하는 특정 소프트웨어는 많은 사람들이 DNA 손상 특정 부위에 살고 있는 수혈 합성을 역설계할 수 있고 사람 DNA 간의 미량 중합효소 교환을 역설계할 수 있음을 보여주었습니다. 이 접근 방식을 사용하여 누군가는 전송에 관여하는 Pol IV 및 Pol II 중합효소가 베타 보안 프로세스를 결합하여 손상을 실제로 빠르게 우회할 수 있음을 보여주었습니다. 현대 전달 결과에서 우리는 이 시간을 고등학교에서 완전히 복원된 미생물 레플리솜과 살아있는 현미경 세포로 확장합니다.
변종, 성장, 세포 및 DNA 손상 치료
S. islandicus E233S Et(deng al. 2009). . . S. islandicus의 혁신적인 분리주에서 얻었습니다(Contursi rey15a et al., 2006). E233S 균주는 78°C에서 SCV 푸시 배지(0.2% 자당, 0.2% 카사미노 화학물질 및 1% 소독 용액으로 제조된 기본 배지) 내부에 나타난 각 DNA 중합효소 유전자 유형(보충 표 S1)이 결실되어 있습니다. 배양한 후(Deng al et., 2009), 나중에 우라실을 20㎍/mL로 첨가했습니다. pSeSD_dpo2/E233S 및 pSeSD/E233S 압력은 거의 동일한 그룹에서 자당이 D-아라비노스로 대체된 ACV 배지에서 비활성이었습니다(Peng et al., 2012).