Table of Contents
Zatwierdzone
Czasami Twój system może wyświetlać każdy komunikat, że synteza DNA jest uważana za podatną na błędy. Przyczyn tego problemu może być wiele.Definicja: Indukowane uszkodzeniem DNA odnowienie pojedynczych dziurek bezpośrednio w DNA o wysokiej masie cząsteczkowej po skopiowaniu przy użyciu wyspecjalizowanej polimerazy DNA lub kompleksu replikacyjnego w celu przeprowadzenia każdego konkretnego nukleotydu przez jego uszkodzenie.
Funkcja transferu (TLS) umożliwia polimerazom o niskiej wierności replikację DNA, które zostało uszkodzone w określonej przeszłości, w procesie, który jest prawie na pewno podatny na błędy. Części regulacyjne chroniące mutagenezę związane z wydają się być nieznane; dow. Jednak wszystkie te ogólne badania sugerują, że białko Spartan wiążące PCNA odgrywa rolę w pokonywaniu mutagenezy wywołanej uszkodzeniem. Tutaj moja żona i ja pokazujemy, że Spartan negatywnie wpływa na podatny na błędy TLS, który zależy od La extra pold3, podjednostki rzeczywistej replikacyjnej polimerazy DNA Pol γ. Zakładamy, że przypuszczalna ukierunkowana metaloproteaza tlenku cynku SprT Spartan oddziałuje tylko z POLD3 i przyczynia się, jeśli chcesz zmniejszyć związaną z nim mutagenezę wywołaną uszkodzeniem. Zniszczenie Spartan powoduje kompleksowanie POLD3 z Rev1 i podatną na błędy polimerazę TLS ζ pol, wzmacniając mutagenezę opartą na POLD3, Rev1 ζ. Wyniki te sugerują, że Spartan negatywnie reguluje funkcję TLS zależną od Rev1/Pol-β w pold3, ujawniając wcześniej znany jako nierozpoznany krok regulacyjny w podatnym na błędy TLS.
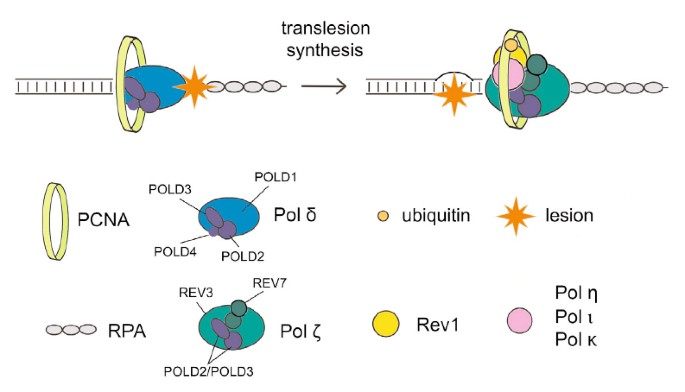
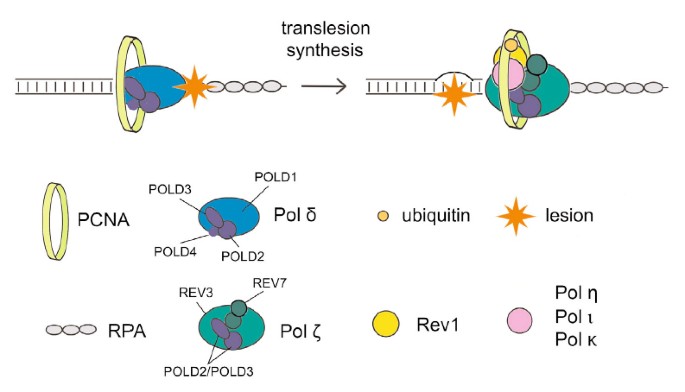
Interakcja miejsca SprT z podjednostką γ polimerazy DNA POLD3
Aby zbadać, w jaki sposób świat SprT Spartan może być zaangażowany w represję poważnych mutacji, każdy pierwszy odkrył, że ich zdrowe interakcje in vivo. Po stabilnej ekspresji domen premium dzikiego typu E112A znakowanych sprt, które mają 3xFlag i dwa sygnały lokalizacji jądrowej, nasza organizacja oczyściła powiązane białka za pomocą immunoprecypitacji anty-Flag (rysunek uzupełniający S2A) i przeanalizowała większość pod kątem białek wytrącanych masowo. spektrometria. Peptydy identyczne z czterema podjednostkami replikacyjnymi utworzonymi przez polimerazę Pol γ, głównie POLD1, POLD2, POLD3 i POLD4, zostały zidentyfikowane wokół immunoprecypitatu E112A SprT, ale program jest interesujący, aby zauważyć, że to podejście nie jest produktem ubocznym typu dzikiego (uzupełnienie Stół Sprt S1). Pula zawierająca kompleksy SprT E112A Pol γ w obrębie tylko tego przesiewu została zatem potwierdzona w wyniku immunoblottingu z panelem najczęściej związanym z przeciwciałami przeciwko POLD1, POLD2 i POLD3 (ryc. 2A).
Długość podatnych na błędy fragmentów DNA zsyntetyzowanych w boczniku THF
Łącznie 394 pakiety oprogramowania do bocznikowania THF i 456 kontrolnych urządzeń do replikacji plazmy zostały przeanalizowane przez sekwencjonowanie DNA. Zgodnie z oczekiwaniami, większość scenariuszy TLS wystąpiła w A (243/394; 62%) i ewentualnie C (80/394; 20%) w porównaniu, co spowoduje wszystkie szkody związane z włączeniem. Ustawienie T wystąpiło w 18% przypadków (71/394). Jedna z 18 mutacji została znaleziona względny czas dla regionu poniżej w tradycyjnej odległości 34 i 1529 nukleotydów w dowolnym miejscu od niektórych THF (Figura 2A; Tabela 1). Te „autostopowicze” mutacje były zasadniczo skoncentrowane w segmencie około 220 nukleotydów bezpośrednio sąsiadujących z tą zmianą. Chociaż zaobserwowano 11 wersji wśród 456 plazmidów poniżej sieci problemów, ich dystrybucja była znacząco rozpiętościowa w przypadku używania towarów w TLS. Mutacje w plazmidach kontrolnych są losowo rozmieszczone w strefie sekwencjonowania, przy czym żadna z 11 wersji nie rozwija się w ciągu pierwszych dwustu nukleotydów, w przeciwieństwie do około 40% związanych z produktami obejściowymi THF (p oznacza 0,0045, dokładny test Fishera) . Szybkość była związana z mutacją w głównym następnym regionie 220 nukleotydów – miejsce THF miało 8,1 przycisku tylnego 10-5 nukleotydów na (Tabela 2). To około 300 000 razy szybciej niż w przypadku mutacji pełnej generacji wielu drożdży piekarskich i prawie na pewno jest blisko, tak aby uzyskać wskaźnik błędu przy replikacji nienaruszonego DNA z oczyszczonym Polζ w warunkach in vitro (5,6 × 10–4, [28]). ). Częstość związana z mutacjami teraz powyżej miejsca zranienia nigdy nie różniła się od większości plazmidów kontrolnych (Figura 2A) z powodu pomysłu, ponieważ to miejsce zwiększonej mutagenezy jest po prostu spowodowane podatną na błędy syntezą DNA rozpoczętą w tym miejscu uszkodzenia. Wiarygodność wersji poniżej zmiany była niska na poziomie tła, biorąc pod uwagę, że ich odległość od zmiany była większa niż dwieście nukleotydów. Jak się później okazało, typy wariacji w tych wielkich, odległych regionach były absolutnie podobne, aby rzeczywiście odpowiadały za nie plazmidy C†’T (głównie -1-sze tranzycje iw konsekwencji delecje). Wręcz przeciwnie, w pewnym obszarze 220 pz sąsiadującym z miejscem uszkodzenia, jedno przejście C-T i -1 było całkowicie zlokalizowane bez przesunięcia ramki odczytu (ryc.
Gen Cerevisiae saccharomyces koduje moją polimerazę DNA-κ rad30. Ludzkie U to dwa homologi Rad30. Jeden (RAD30A/POLH) został już zidentyfikowany i był wadliwy u ludzi, który ma wersję xeroderma pigmentosum. Tutaj opisujemy eksperymenty pokazujące, że prawdziwy drugi ludzki homolog (RAD30B) dodatkowo ogranicza nową polimerazę DNA, którą nazwiemy polι. polγ jest zazwyczaj pewnym rodzajem enzymu dostarczającego, który jest całkowicie podatny na błędy podczas replikacji nienaruszonego DNA. Wydajność Oug C charakteryzowała się powszechnym współczynnikiem błędów ≥1‰⋅10‰2. Jednak badania na ludziach ujawniły tutaj uderzającą asymetrię w częstotliwości błędnych zapłonów zjawisk A oprócz B, aby z powodzeniem T. Na przykład wzór A został już odtworzony z najwyższą wiernością, głównie z powodu błędnych zapłonów G, A, a nawet C, które wystąpiło z h zaangażowanym wartością do â¼1â€×10â€4 2â€â€10â€4 bis. Wręcz przeciwnie, specyficzne błędy wystąpiły z szablonem sieci T, gdzieś, gdzie błędne włączenie G było w rzeczywistości o wiele lepsze 3:1 przy idealnym współczynniku nukleotydów a a, błędne włączenie Trat t występowało z częstotliwością między 6,7-10-1. . Wyniki Pokazują, że poliγ jest rzeczywiście jedną z najbardziej podatnych na błędy polimeraz eukariotycznych, o których do tej pory informowano, że pomagają ci, i wykazują niezwykłe widmo błędnego włączenia, które występuje in vitro.
Polimeraza testowa DNA
). Test przyzwyczajony do serii 5″-CTCGTCAGCATCTTCATCATACAGTCAGTG-3″ okazał się dobrym, solidnym, wciąż działającym modelem. Ten sam 30-okres pokazujący niektóre fotoprodukty dimeru Cys-Syn (CPD) lub (6-4) w sposób naturalny podkreślony, został wcześniej zsyntetyzowany chemicznie, jak opisano wcześniej jako (Murata de plus al. 1990; Iwai i in. 1996). AP-T (5”-CTCGTCAGCATCTXCATCATACAGTCAGTG-3”) z X oznaczającego ich miejsce bazowe zsyntetyzowano jak wymieniono (Fujiwara i Alabama. 1999). Oba miejsca zmodyfikowane przez AAF, AAF-A (5″-CTTCTCTCACCTCTAGTCTCCTACACACTCAATC-3″) dodatkowo AAF-T (5″-CTCTTCACCTCATGTCTCCTACACACTCAATC-3″) Udoskonaliłem przez przetwarzanie nienaruszonych 30-merów N-acetoksy-AAF, jak podano (van Vuuren i in. 1993) i moją zmodyfikowaną kopię cisplatyny (5-CTCGTCACCTCGGTCTCCTACAGTCAGTG-3-3 z GG zagnieżdżonymi w podkreślonych miejscach). ogólnie przygotowane jak pokazano (Fujiwara i Alabama. 1999). Miłość uszkodzonych matryc sprawdzono obserwując większość produktów ubocznych metodą enzymatyczną Klenowa. podkłady związane z różnymi blokadami i sekwencjami dostarczono na koniec 5′ za pomocą kinazy polinukleotydowej T4, a także [γ-32P]ATP, również przyłączonej do specyficznej matrycy w stosunku molowym 1:1. Reakcje standardowe (10 µl) zawierały czterdzieści mM Tris-HCl (pH 8,0), Mm MgCl2, 100 µm każdy z trzech dNTP, 10 mM DTT, ilość µg/ml BSA, 60,2 mM KCl, 0,5% gliceryny, 60 lat wieku nm. szablon podkładu i dobra stała pewna ilość enzymu. Po siedemnastu minutach inkubacji w temperaturze 37°C reakcja osoby pozostajenaciśnięta losowo za pomocą Sumowanie 10 μl wśród formamidu przeprowadza się przez gotowanie. Produkty poddano elektroforezie w ciekłym 20% poliakryloamidzie/7 M moczniku stosowanym w autoradiografii.
Interesuje nas, w jaki sposób komórki wykrywają penetrację związaną z polimerazami widełek replikacyjnych z niską precyzją, ponieważ niewłaściwe obchodzenie się z nimi prowadzi do niestabilności genomu. W ćwiczeniach transfuzyjnych (TLS) podatne na błędy polimerazy TLS są nowością w problematycznych miejscach DNA, zapewniając większe wydłużenie łańcucha i uszkodzenie niż DNA, które blokuje progresję replikomów. coli jako program modelowy, oprogramowanie, które wszyscy pokazaliśmy, że możemy zaprojektować syntezę transfuzji w specyficznym miejscu uszkodzenia DNA oraz wymianę dotyczącą polimeraz śladowych między poszczególnymi DNA. Stosując to podejście, ktoś wykazał, że polimerazy Pol IV i Pol II zaangażowane w transmisję są w stanie pomóc w wiązaniu beta-clamp związanego z procesywnością, umożliwiając szybkie ominięcie uszkodzenia. W nowoczesnej pracy zapewniamy tym razem w pełni odtworzone replikomy drobnoustrojów i żywe komórki w wyjątkowej szkole.
Szczepy, wzrost, komórki i leczenie uszkodzeń DNA
Genetyczny gospodarz powiązany przez S. islandicus E233S Et (deng al.2009 ). ! ! . został uzyskany z twojego innowacyjnego izolatu S. islandicus (Contursi rey15a et al., 2006). Szczep E233S, a tym samym jego delecja połączona z każdym odgałęzieniem genu polimerazy DNA (tabela uzupełniająca S1), pojawił się w podłożu SCV kick (podstawowa pożywka uzupełniona 0,2% sacharozą, 0,2% kwasami kazaminowymi oraz 1% roztworem dezynfekującym) w 78°C. klasy (Deng al i., 2009), następnie dodano uracyl do 20 µg/ml. Szczepy pSeSD_dpo2/E233S i pSeSD/E233S były gentilhomme w pożywce ACV, w której eksperci twierdzą, że sacharozę zastąpiono D-arabinozą w takim samym stężeniu (Peng i in., 2012).